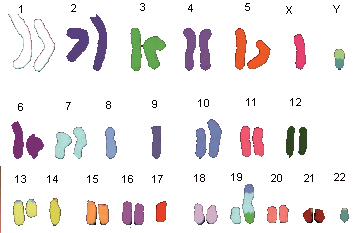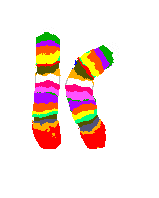Ausgangslage:
1. Man kennt ein großes Molekül (z.B. Protein, Enzym, Hormon, Rezeptor, DNS, RNS), man kennt seinen Aufbau oder zumindest einen wesentlichen und charakteristischen Teil davon.
2. Man hat ein Untersuchungsmaterial nämlich ein Molekülgemisch in Lösung, oder das Molekülgemisch einer lebenden oder toten Zelle oder ein lebendes oder totes Gewebe oder eine undefinierte Mischung aus allem.
3. Man hat folgende Fragen: Existiert zumindest eines der gesuchten großen Moleküle oder Molekülgruppen im Untersuchungsmaterial ? Wenn ja, wo liegt es im Untersuchungsmaterial ? Wenn mehr als eines exisitiert, wieviele sind es und wo sind sie konzentriert ?
4. Problem: Es gibt keine Mikroskope oder sonstigen Geräte, mit denen man Moleküle so genau ansehen kann, dass man das gesuchte entdecken könnte. Das Problem ist weniger die Ortsauflösung (mit Rastertunnelmikroskopen könnte man einzelne Atome sehen) sondern es fehlt die Selektivität. Es ist unmöglich ein bestimmtes Molekül unter Millionen ähnlicher Moleküle zu finden.
5. Forschungsmotivation: In der Human-, Tier-, Pflanzen- und Zytogenetik will man wissen, auf welchen Chromosomen und an welcher Stelle bestimmte Gene oder Gen-Gruppen lokalisiert sind und wie deren Einbau ins menschliche Genom verlaufen ist.
6. Diagnostische Motivation: In der Histo- und Zytopathologie benötigt man enorme Spezifität/Selektivität um das Vorhandensein von AIDS-, Heptitis-, Bakterien-DNS und -RNS in Schnittpräparaten und Abstrichen nachzuweisen.
Lösung:
Man baut synthetisch ein Markermolekül, das sich an das gesuchtes Molekül frei- und bereitwillig andockt nach dem Schlüssel-Schloß-Prinzip nach dem Vorbild der bekannten Antigen-Antikörper-Reaktion aus der Immunzytologie. Dieses Markermolekül dient als chemische Sonde.
Das Markermolekül bekommt einen molekularen Ballast, der es weithin sichtbar macht. Dieser Ballast kann ein radioaktiver Strahler (Radiomarker), ein Lichtstrahler (Photomarker) oder ein Fluoreszenzstrahler (Fluoreszenzmarker=Fluorochrome) sein. Die Zukunft gehört den Fluorochromen.
Die Herstellung solcher Sonden ist schwer. Man muss das gesuchte Molekül so gut kennen, dass man eine chemische Komplementärstruktur herstellen kann, die zum gesuchten wie ein Schlüssel in ein Schloß passt.
Der Schlüssel darf nicht in mehrere Schlösser passen, er muss möglichst hochselektiv für das gesuchte Molekül sein. Er muß in Lösung oder im Gewebe zumindest eine gewisse Zeit beständig und beweglich sein. Wenn er im Zellinnenraum wirksam sein soll, muss er die Zellmembran überwinden können.
Ist ein solches Komplementärmolekül gefunden oder systematisch synthetisiert, muss es nun mit einem markertragenden Ballastteil so gekoppelt werden, dass der Ballast den Schlüssel-Schloßmechanismus nicht stört und trotzdem weithin sichtbar bleibt. Schließlich muß ein Verfahren gefunden werden, mit dem man das komplette Markermolekül massenhaft herstellen kann.
Zwei Schlüssel-Schloß-Mechanismen sind z.Z. biochemisch verfügbar:
(1) Zum Auffinden von DNA/RNA benutzt man Sonden-DNA/RNA mit komplementärer Basenpaarung.
(2) Zum Auffinden von Proteinen benutzt man Antikörper.
|
Viele Körper strahlen Licht und Wärme ab, wenn man ihnen vorher Energie zuführt.
Strahlt der Körper nur Licht ab ohne Wärmeabstrahlung (was nur wenige Körper tun), dann spricht man von Luminiszenz. Man unterscheidet zwei Arten von Luminiszenz.
1. Phosphoreszenz, wenn ein langdauerndes Nachleuchten auftritt (Anwendung: Speicherfolien).
2. Fluoreszenz, wenn das Leuchten sofort nach Energiezufuhr (=Stimulierung) einsetzt und nur kurz anhält (spontane Emission bis ca. 10-3 sec).
Bei Fluoreszenz werden Elektronen durch hochenergetisches kurzwelliges ultraviolettes Licht aus dem Valenzband ins Leitungsband gestoßen. Man braucht je Elektron-Loch-Paar ungefähr das Dreifache der Energie des Bandabstandes. Sofort danach rekombinieren die Elektronen mit einem Loch unter Aussendung von Licht, das in der Regel niederenergetischer ist als der Stimulus. Die einzelnen Fluoreszenzfarbstoffe geben je eine charakteristische Lichtantwort im sichtbaren oder infraroten Spektralbereich. Man kann gleichzeitig mehrere oder viele Marker einsetzen und damit gleichzeitig nach mehreren oder vielen Substanzen fahnden. Die Farben charakterisieren die Markersubstanzen und damit sekundär die Strukturen, an denen diese angedockt sind.
|
1970 entdeckte T. Caspersson einen für Nucleobasen selektiven Fluoreszenzfarbstoff, das Quinacrin. Damit gefärbt zeigt jedes Chromosom ein spezifisches Muster von Querstreifen, das sogenannte Q-Bandenmuster. Durch diese Banden konnte man zum ersten Mal alle 22 Autosomenpaare und die zwei Geschlechtschromosomen des Menschen unterscheiden. Sumner, Schnedl, Seabright entdeckten bis 1971 weitere Farbstoffe und Präparationsmethoden, die neue Bandenmuster erzeugen: G-, R-, T-, C-Banden. Dunkle Banden enstehen dort, wo DNS dichter gepackt ist, helle dort wo die DNS lockerer gepackt vorliegt. Außerdem beeinflußt das lokale Mengenverhältnis der Basenpaare A-T zu G-C die Bandenbildung. Jedenfalls darf man nicht meinen, dass eine Bande einem Gen oder einer Gengruppe funktionell entspräche. Banden haben keine Funktion, sie sind Erkennungsmerkmale für relativ grobe Organisationsschemata von Chromosomen.
- Caspersson,T, et al.: Differential banding of alkylating fluorochromes in human chromosomes. Exp. Cell Res. 60 (1970) 315-319
- Sumner,A., et al.: New technique for distinguishing between human chromosomes. Nature 232 (1971) 31-32
- Schnedl, W.: Analysis of the human karyotype using a reassociation technique. Chromosoma 34 (1971) 448-454
- Seabright,M.: A rapid banding technique for human chromosomes. Lancet II (1971), 971-972.
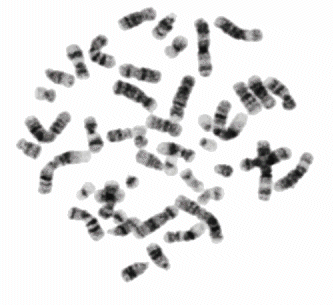
Chromosomen in GTG-Färbung
 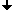
|
Wie kommt die Bezeichnung "Fluoreszenz in situ Hybridisierung" zustande ?
Der lateinische Ausdruck "in situ" ist eine Lagebezeichnung. Sie bedeutet am natürlichen Ort, in üblicher Lage, so wie das Ding ohne Eingriff eines Wissenschaftlers daliegt. Der Gegenbegriff heißt "in vitro". Er bedeutet am unnatürlichen, künstlich umgepflanzten Ort, im Reagenzglas.
Obwohl sich bei FISH die Chromosomen keineswegs an einem natürlichen Ort befinden (nämlich in einem Zellkern im Inneren einer Zelle im Inneren eines Menschen), sondern auf einem Objektträger, bezeichnet der Genetiker trotzdem diesen Platz aus seiner Sicht als "in situ", nämlich als natürlichen Aufenthaltsort für Chromosome.
Das lateinische Wort "hybrida" bedeutet Bastard, Mischling.
Ungepaarte Basenstränge völlig verschiedener Herkunft können spontan hybridisieren, falls sie wie Schlüssel und Schloß zusammenpassen.
Die Grundidee von FISH ist: Hat man unbekannte DNA, dann nehme man bekannte DNA (Sonde) und hybridisiere beide. Überall dort, wo eine Bindung stattfindet ist die unbekannte DNA enthüllt, sie ist schlicht komplementär identisch mit der gebundenen Sonde.
Die Schwierigkeit liegt in der Herstellung spezifischer DNA-Sonden:
Man muss spezifische DNA-Fragmente (ca. 200 Femtogramm) aus langen Strängen isolieren. Man fragmentiert durch strangbrechende Enzyme (z.B. Endonuclease DNase I, Nick-Translation) oder mechanisch durch Sezieren eines Chromosoms mit einer feinen Glasnadel. Nach sorgfältiger Neutralisierung der übrigbleibenden unspezifische DNA vervielfältigt man das spezifische Fragment mit Hilfe der DOP-Polymerase-Kettenreaktion und baut dabei in jedes Stück mindestens einen Fluoreszenzmarker ein. Dann trennt man die komplementären Basenstränge auf und erhält damit eine beträchtliche Menge einer brauchbaren DNA-Sonde.
Man spricht von einer DNA-Bibliothek, wenn man mehrere/viele verschiedene solcher Sonden von wohldefinierten Chromosomenabschnitten hergestellt oder gekauft hat.
Besitzt man solche Sonden, kann man unbekanntes Chromosomenmaterial folgendermassen kartieren:
Man muss Sonden und Targets zunächst vorbehandeln (Basenstränge auftrennen, Verunreinigungen entfernen, denaturieren). Man gibt überschüssig viel Material einer Sonde (oder damit es schneller geht gleichzeiig überschüssig viel Material mehrerer verschiedener Sonden) zum Target hinzu und stellt Zeit und Umgebungsbedingungen bereit, dass Hybridisierung stattfinden kann ="in situ Hybridisierung". Danach wäscht man sorgfältig das überschüssige Sondenmaterial aus, bis man sicher ist, dass keine freien, nichthybridisierten Sonden zurückbleiben.
Das Ergebnis ist ein DNA-Hybrid bestehend je zur Hälfte aus unbekannter DNA und wohlbekanntem Sondenmaterial. Unter dem Fluoreszenzmikroskop bringt man die Sonden zum Leuchten und erhält je ein Farbsignal dort, wo die Sonden angedockt haben. Man kartiert die Positionen der Farbsignale am unbekannten Chromosom und vergleicht mit bekannten Karten.
Die Pioniere der Fluoreszenz in Situ Hybridisierung waren:
- Langer, PR., et al.: Enzymatic synthesis of Biotin-labeled polynucleotides. Proc. Natl. Acad. Sci. USA 78 (1981), 6633-6637
- Pinkel, D., et al.: Cytogenetic analysis using quantitative, high sensitivity fluorescence hybridization. Proc. Natl. Acad. Sci. USA 83 (1986), 2934-2938
|
Um 24 Chromosomen unterscheiden zu können braucht man zwar mindestens 24 Sonden aber es genügen 6 Farbstoffe. Man braucht einen Farbstoff, der alle Chromosomen gemeinsam färbt und Banden erzeugt, damit man die Chromosomen an diesen Banden unterscheiden kann (DAPI blau = Gegenfärbung). Die restlichen 5 Farbstoffe muss man so auf die Sonden aufteilen, dass jedes Chromosom eine charakteristische Farbkombination erhält.
Erstpublikationen:
Nederlof, P., et al.: Multip. fluorescence in situ hybridisation. Cytometry 11 (1990), 126-131.
Schröck, E., et al.: Multicolor spectral karyotyping of human chromosomes. Science 273 (1996), 494-497.
Speicher, M., et al.: Karyotyping human chromosomes by combinatorial multi-color FISH. Nature Genet. 12 (1996) 368-375.

|
Färbetabelle:
Die 5 Spalten enthalten die 5 Farbstoffe
Die 24 Zeilen bezeichnen die menschlichen Chromosomen
Spectrum OrangeTM is a trademark of Vysis, Inc.
Texas Red® is a registered trademark of Molecular Probes, Inc.
CyTM5 and Cy™ are trademarks of Amersham Pharmacia Biotech Ltd., Inc.
Mit 6 schmalbandigen optischen Filtern auf einem motorgesteuerten Filterrad werden am Fluoreszenzmikroskop (Software isis/mFish mit Axioplan 2 mot) 6 digitale Fluoreszenzbilder aufgenommen. Jedes zeigt nur einen Farbstoff. Die 6 deckungsgleichen Monofarb-Bilder werden zu einem vereinigt (jedes Pixel besitzt nun 6 Farbmesswerte a 8 Bit) und gespeichert mit 6x8 = 48 Bit/Pixel.
Die 6 Messwerte eines Pixels bilden einen Punkt in einem 6D-Farbraum und dieser lässt sich auffassen als die 6-Kanal-Echtfarbe des Pixels.
Solche Echtfarben kann man jedoch nicht auf RGB-Farben abbilden und auf Graphikkarten realisieren. Man verwandelt deshalb die Echtfarben in exakt 24 willkürliche künstliche Falschfarben, je eine pro Chromosom, die sogenannten Klassifizierungsfarben. Dann richtet man die Chromosomen senkrecht aus und ordnet sie nacheinander in Zeilen an zum Karyogramm.
isis/mFish is a trademark of MetaSystems GmbH, Altlussheim.
Axioplan 2 mot is a registered trademark of Carl Zeiss, Jena.
|
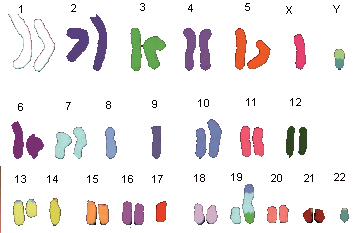
|
Falschfarben-mFish-Karyogramm
Beispiele für willkürliche Falschfarben:
Cy5 von Chromosom 1 codiert als Weiss.
DEAC von Chromosom 2 codiert als Magenta.
TexRed von Chromosom 3 codiert als Blassgrün.
FITC+SpeOra von Chromosom 12 codiert als Olivgrün.
Das Karyogramm zeigt einen schwer pathologischen Chromosomensatz einer männlichen Tumorzelle (Adenom HRT 18).
Unmittelbar sichtbare grobe Defekte: Die Chromosomen 8, 9, 14, 17, 22 gibt es nur einfach. Chromosom 19 enthält Teile von 10 und 3 und Chromosom Y enthält das fehlende Chromosom 22.
|
Mit geeigneter Software kann man aus der 6-Kanal-Echtfarbe jedes einzelnen Pixels dessen Zugehörigkeit zu einem Chromosom klassifizieren lassen. Das bedeutet, dass kleine Translokationen von Genmaterial von einem Chromosom auf ein anderes detektierbar sind.
Man kann einzelne Chromosomen mikrosezieren (man braucht 5 bis 10 überlappende Abschnitte pro Chromosom) und daraus regionen-spezifische DNA-Bibliotheken herstellen, aus denen man Sonden vervielfältigt. Diese Sonden färben nur einen bestimmten Teil ihres Chromosoms.
Erstpublikationen:
Senger, G., et al.: Microdissection of banded human chromosomes. Hum. Genet. 12 (1990), 507-511.
Telenius, H., et al.: Degenerate oligonucleotide-primed PCR. Genomics 13 (1992), 718-725.
Chudoba, I., et al.: High resolution multicolor banding: a new technique for refined FISH analysis of human chromosomes. Cytogenet. Cell Genet. 84 (1999), 156-160.

Schema der PCP-Färbung (Partial Chromosome Paint) der Chromosmen 1, 5 und 7
nach I. Chudoba mit 8 überlappenden regionalen Sonden.
Chromosom 5 in PCP-Färbung nach I. Chudoba
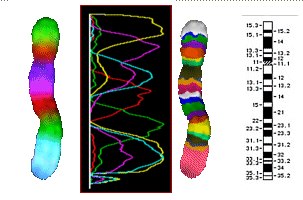
| oberes Bild links: | additive Farbmischung der 8 Sonden |
| oberes Bild halblinks: | die 8 überlagerten Fluorezenzamplituden der 8 Sonden |
| oberes Bild halbrechts und Bild unten: | Falschfarben so, dass sich möglichst signifikante Bänder zeigen |
| oberes Bild rechts: | bekannte Banden von Chromosom 5 |
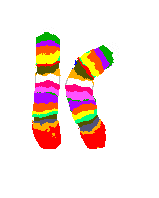
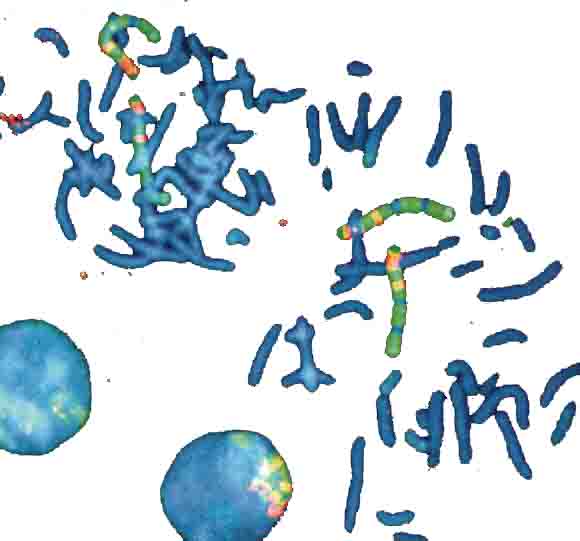
4 Chromsomensätze, davon 2 lose in Metaphase und 2 gepackt in 2 Zellkernen.
DAPI-blau-Färbung zeigt unspezifisch alle Chromosomen.
4 Paare von Chromosom 1 (im linken Zellkern kaum zu sehen, weil verdeckt von anderen Chromosomen) in charakteristischer Multicolor-Bänderung.
Aufnahme V. Beensen, FSU Jena, Inst. f. Humangenetik
aus Innovation Lambda4 Carl Zeiss Magazine of Microscopic Imaging and Analysis, 6/2000.
 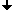
|
DNS und RNS sind riesige Kettenmoleküle, die unter bestimmten Umständen in stark geknäulter Form als Chromosomen sichtbar sind. Der Code ist für Viren, Bakterien, pflanzliche, tierische und menschliche Zellen identisch. Er basiert auf 4 Buchstaben, den 4 wichtigen Nucleotiden, die sich jeweils durch das zentrale Basenmolekül unterscheiden (Purin oder Pyrimidin: Adenin/A, Thymidin/T, Guanin/G und Cytosin/C). Die Nucleotide bilden enorm lange z.T. gestreckte, z.T. verknäulte Ketten. Eine solche Kette nennt man Basensequenz und misst deren Länge in der Einheit kB (=Kilobasen nicht Kilobyte!).
Ca. 5 Prozent der Basensequenz kodieren Information zum Bau von Proteinen oder komplexe Steuerinformation (z.B. Körperbau). Das Ablesen basiert wiederum auf dem Schlüssel-Schloß-Prinzip. Es lagern sich jeweils 2 komplementäre Nucleotide (A-T und C-G) aneinander, d.h. eine Basensequenz in der DNS von CCATCAA wird exakt von einer komplementären Basensequenz GGTAGTT abgelesen.
top of page: |